教員個人(研究室)ページ
ゲノム機能工学研究室
Laboratory of Genomic Function Engineering

<研究略歴>
1999年東京大学大学院農学生命科学研究科応用動物科学専攻博士課程修了。1999年日本学術振興会特別研究員(PD)、東京大学大学院助手、2007年より同大学院助教としてエピジェネティクス研究に従事。
その間2003-2006年トロントMt Sinai Hospital (Dr Andras Nagy Lab)博士研究員として幹細胞生物学およびゲノム改変研究手法を習得。
2011年明治大学農学部・専任講師、2014年同・専任准教授を経て、2019年より現職。
<主な担当科目>
遺伝学、ゲノム機能工学、生命科学実験II<ひとこと>
生物が本来持っているゲノム機能を制御して、有用細胞・個体や病態モデル動物の作出につながる研究を楽しもう!研究テーマ
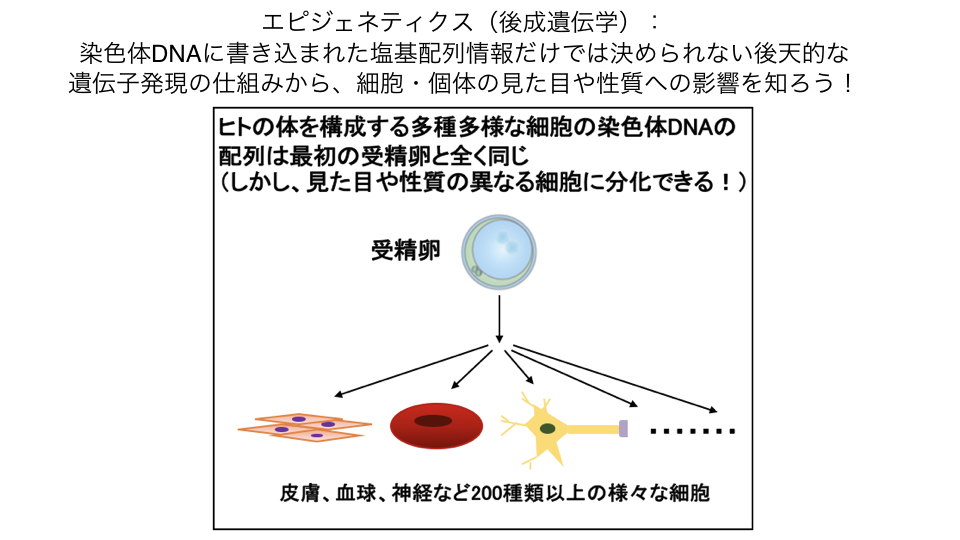
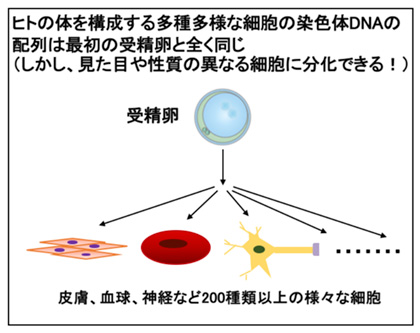
当研究室では、ゲノムDNAのメチル化やヒストン修飾による後天的な発現制御であるエピジェネティクスについて、組織・細胞種特異的なDNAメチル化を中心に研究をしています。
私たちの体は、形態や機能の異なる200種ほどの細胞種で構成されています。
一方、約200種の細胞は、ゲノムDNAにコードされた全く同じ遺伝情報を設計図として、発生に伴って出来上がってきます。
したがって、各細胞種は必要な遺伝子と不必要な遺伝子のオン・オフを厳密に制御することで正しく形成されると考えられます。
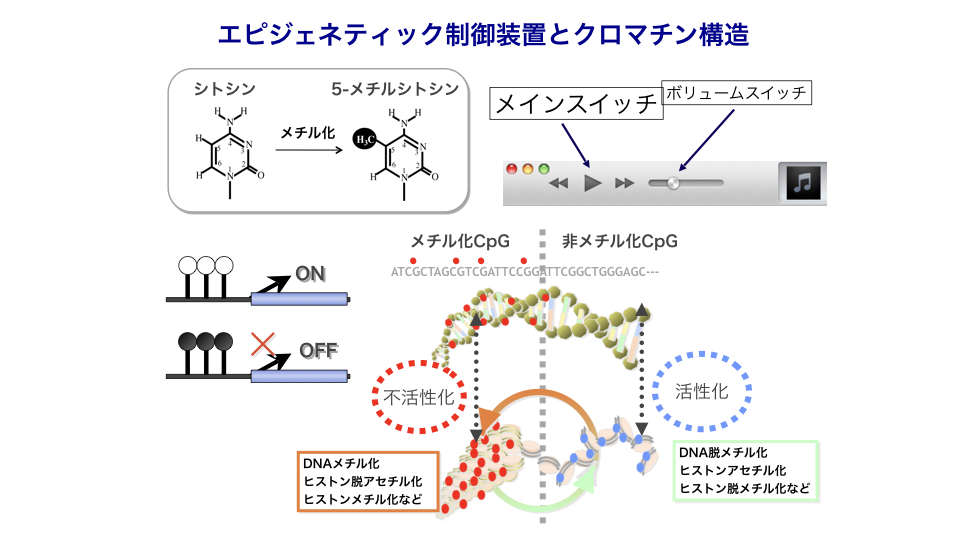
DNAメチル化は不必要な遺伝子の働きを完全にオフにする遺伝子発現のメインスイッチとして働いています。
体細胞核移植クローン動物の作出、iPS細胞の樹立成功などが大きな転機となり、同一の遺伝情報から異なる細胞種で必要とされる遺伝子セットを効率的に発現させる(させない)ための根幹メカニズムとしてエピジェネティクスが注目されており、高速次世代シーケンサーなども積極的に活用しながら、組織・細胞種毎のエピゲノム制御を同定できています。
さらに、再生医療に用いられる多能性幹細胞や有用細胞が人為的なエピジェネティック制御により、効率よく樹立できる可能性があります。 また、組織特異的なエピジェネティック制御は正常な発生にも重要ですが、逆にその破綻は病気の原因となるとも考えられています。 現在では多くの慢性疾患はDNA塩基配列の変化ではなく、特定遺伝子のエピジェネティック異常の蓄積により発症することが示唆されています。 このことから、細胞や組織の機能発現に重要な遺伝子のエピジェネティック異常を誘発することで新たな疾患モデル作出につながる可能性もあります。 現在は、特定の遺伝子のみに限定したエピゲノム改変により、従来の遺伝子操作とは異なるゲノム機能改変を目指しています。今後は、げっ歯類のみならず、ヒトにより近いブタでの有用細胞や病態モデル動物の新たな作出法の確立を進めたいと思っています。
研究室メンバー
修士2年 0名; 修士1年 2名; 学部4年 8名; 学部3年 9名; 助教 1名研究業績
2011年以降
W Wang, T Ito, S Otsuka, H Nansai, K Abe, Y Nakao, J Ohgane, M Yoneda, H Sone. Epigenetic effects of insecticides on early differentiation of mouse embryonic stem cells. Toxicol In Vitro. 75, 105174, 2021.
Y Arai, K Umeyama, N Okazaki, K Nakano, K Nishino, H Nagashima, J Ohgane* (Correspondence author). DNA methylation ambiguity in the Fibrillin-1 (FBN1) CpG island shore possibly involved in Marfan syndrome. Sci Rep. 10, 5287, 2020.
N Haga, M Kobayashi, N Michiki, T Takano, F Baba, K Kobayashi, H Ohyanagi, J Ohgane, K Yano, K Yamane. Complete chloroplast genome sequence and phylogenetic analysis of wasabi (Eutrema japonicum) and its relatives. Sci Rep. 9, 4377, 2019.
R Maekawa, R Ito, Y Iwasaki, K Saito, K Akutsu, S Takatori, R Ishii, F Kondo, Y Arai, J Ohgane, K Shiota, T Makino, N Sugino. Evidence of exposure to chemicals and heavy metals during pregnancy in Japanese women. Reprod. Med. Biol. 16, 337-348, 2017.
Y Arai, K Umeyama, K Takeuchi, N Okazaki, N Hichiwa, S Yashima, K Nakano, H Nagashima, J Ohgane* (Correspondence author). Establishment of DNA methylation patterns of the Fibrillin1 (FBN1) gene in porcine embryos and tissues. J. Reprod. Dev. 63, 157-165, 2017.
H Nishihara, S Yoshida, N Kanno, N Nishimura, H Ueharu, J Ohgane, T Kato, Y Kato. Involvement of DNA methylation in regulating rat Prop1 gene expression during pituitary organogenesis. J. Reprod. Dev. 63, 37-44, 2017.
Y Arai, H Fukukawa, T Atozi, S Matsumoto, Y Hanazono, H Nagashima, J Ohgane* (Correspondence author). Ultra-Deep Bisulfite Sequencing to Detect Specific DNA Methylation Patterns of Minor Cell Types in Heterogeneous Cell Populations: An Example of the Pituitary Tissue. PLoS One. 11, e0146498, 2016.
Y Arai, K Hayakawa, D Arai, R Ito, Y Iwasaki, K Saito, K Akutsu, S Takatori, R Ishii, R Hayashi, S Izumi, N Sugino, F Kondo, M Horie, H Nakazawa, T Makino, M Hirosawa, K Shiota*, J
Ohgane*. (*Co-correspondence authors) Putative Epimutagens in Maternal Peripheral and Cord Blood Samples Identified Using Human Induced Pluripotent Stem Cells. Biomed. Res. Int. 2015, 876047, 2015.
D Arai, K Hayakawa, J Ohgane, M Hirosawa, Y Nakao, S Tanaka, K Shiota. An epigenetic regulatory element of the Nodal gene in the mouse and human genomes. Mech. Dev. 136, 143-154, 2015.
Y Arai, J Ohgane*, S Fujishiro, K Nakano, H Matsunari, M Watanabe, K Umeyama, D Azuma, N Uchida, N Sakamoto, T Makino, S Yagi, K Shiota, Y Hanazono, H Nagashima*. (*Co-correspondence authors) DNA methylation profiles provide a viable index for porcine pluripotent stem cells. Genesis. 51, 763-776, 2013.
K Hayakawa, J Ohgane, S Tanaka, S Yagi, K Shiota. Oocyte-specific linker histone H1foo is an epigenomic modulator that decondenses chromatin and impairs pluripotency. Epigenetics. 7, 1029-1036, 2012.
K Hayakawa, MO Nakanishi, J Ohgane, S Tanaka, M Hirosawa, MJ Soares, S Yagi, K Shiota. Bridging sequence diversity and tissue-specific expression by DNA methylation in genes of the mouse prolactin superfamily. Mamm. Genome. 23, 336-345, 2012.
R Maekawa, S Yagi, J Ohgane, Y Yamagata, H Asada, I Tamura, N Sugino, K Shiota. Disease-dependent differently methylated regions (D-DMRs) of DNA are enriched on the X chromosome in uterine leiomyoma. J Reprod Dev. 57: 604-12, 2011.
Y Arai, J Ohgane, S Yagi, R Ito, Y Iwasaki, K Saito, K Akutsu, S Takatori, R Ishii, R Hayashi, SI Izumi, N Sugino, F Kondo, M Horie, H Nakazawa, T Makino, K Shiota. Epigenetic Assessment of Environmental Chemicals Detected in Maternal Peripheral and Cord Blood Samples. J Reprod Dev 57: 507-517, 2011.
2010年以前(主要な業績のみ)
H Muramoto, S Yagi, K Hirabayashi, S Sato, J Ohgane, S Tanaka, K Shiota. Enrichment of short interspersed transposable elements to embryonic stem cell-specific hypomethylated gene regions. Genes Cells 15: 855-865, 2010.
S Sato, S Yagi, Y Arai, K Hirabayashi, N Hattori, M Iwatani, K Okita, J Ohgane, S Tanaka,T Wakayama, S Yamanaka, K Shiota. Genome-wide DNA methylation profile of tissue-dependent and differentially methylated regions (T-DMRs) residing in mouse pluripotent stem cells. Genes Cells. 15: 607-618, 2010
M Oda, S Tanaka, Y Yamazaki, H Ohta, M Iwatani, M Suzuki, J Ohgane, N Hattori, R Yanagimachi, T Wakayama, K Shiota. Establishment of trophoblast stem cell lines from somatic cell nuclear-transferred embryos. Proc Natl Acad Sci USA. 106: 16293-16297, 2009
K Sato, H Hukata, Y Kogo, J Ohgane, K Shiota and C Mori. Neonatal exposure to diethyl-
stilbestrol alters the expression of DNA methyltransferases and methylation of genomic DNA in the mouse uterus. Endocr J. 56: 131-139, 2009
S Yagi, K Hirabayashi, S Sato, W Li, Y Takahashi, T Hirakawa, G Wu, N Hattori, J Ohgane, S Tanaka, XS Liu, K Shiota. DNA methylation profile of tissue-dependent and differentially methylated regions (T-DMRs) in mouse promoter regions demonstrating tissue-specific gene expression. Genome Res. 18: 1969-1978, 2008
H Asada, Y Yamagata, T Taketani, A Matsuoka, H Tamura, N Hattori, J Ohgane, N Hattori, K Shiota, N Sugino. Potential link between estrogen receptor-a gene hypomethylation and uterine fibroid formation. Mol Hum Reprod. 14: 539-545, 2008
H Sakamoto,Y Kogo, J Ohgane, N Hattori, S Yagi, S Tanaka, K Shiota. Sequential changes in genome-wide DNA methylation status during adipocyte differentiation. Biochem Biophys Res Comm. 366: 360-366, 2008
S Senda,T Wakayama, Y Arai, Y Yamazaki, J Ohgane, S Tanaka, N Hattori, S Yagi, R Yanagimachi, K Shiota. DNA methylation errors in cloned mice disappear with advancement of aging. Cloning Stem Cells. 9: 293-302, 2007
N Hattori, Y Imao, K Nishino, N Hattori, J Ohgane, S Yagi, S Tanaka, K Shiota. Epigenetic regulation of Nanog gene in embryonic stem and trophoblast stem cells. Genes Cells. 12: 387-396, 2007
K Sato, H Fukada, Y Kogo, J Ohgane, K Shiota, C Mori. Neonatal exposure to diethyl- stilbestrol alters the expression of DNA methyltransferases and methylation of genomic DNA in the epididymis of mice. Endocr J. 53: 331-337, 2006
J Ohgane, T Wakayama, S Senda, Y Yamazaki, K Inoue, A Ogura, J Marh, S Tanaka, R Yanagimachi, K Shiota. The Sall3 locus is an epigenetic hotspot of aberrant DNA methylation associated with placentomegaly of cloned mice. Genes Cells. 9: 253-260, 2004
T Imamura, S Yamamoto, J Ohgane, N Hattori, S Tanaka, K Shiota. Non-coding RNA directed DNA demethylation of Sphk1 CpG island. Biochem Biophys Res Comm. 322: 593-600, 2004
S Senda, T Wakayama, Y Yamazaki, J Ohgane, N Hattori, S Tanaka, R Yanagimachi, K Shiota. Skewed X-inactivation in cloned mice. Biochem Biophys Res Comm. 321: 38-44, 2004
U Singh, L Fohn, T Wakayama, J Ohgane, C Steinhoff, B Lipkowitz, R Schulz, A Orth, H Ropers, R Behringer, S Tanaka, K Shiota, R Yanagimachi, U Nuber, R Fundele. Different molecular mechanisms underlie placental overgrowth phenotypes caused by interspecies hybridization, cloning, and Esx1 mutation. Dev Dyn. 230: 149-164, 2004
N Hattori, K Nishino, Y Ko, N Hattori, J Ohgane, S Tanaka, K Shiota. Epigenetic control of mouse Oct-4 gene expression in embryonic stem cells and trophoblast stem cells. J Biol Chem. 279: 17063-17069, 2004
J Ohgane, N Hattori, M Oda, S Tanaka, K Shiota. Differentiation of trophoblast lineage is associated with DNA methylation and demethylation. Biochem Biophys Res Comm. 290:701-706, 2002
K Shiota,Y Kogo, J Ohgane, T Imamura, A Urano, K Nishino, S Tanaka, N Hattori. Epigenetic marks by DNA methylation specific to stem, germ and somatic cells in mice. Genes Cells 7: 961-969, 2002.
J Ohgane, T Wakayama, Y Kogo, S Senda, N Hattori, S Tanaka, R Yanagimachi, K Shiota. DNA methylation variation in cloned mice. Genesis. 30: 45-50, 2001
J Cho, H Kimura, T Minami, J Ohgane, N Hattori, S Tanaka, K Shiota. DNA methylation regulates placental lactogen I gene expression. Endocrinology. 142: 3389-3396, 2001
T Imamura, J Ohgane, S Ito, T Ogawa, N Hattori, S Tanaka, K Shiota. CpG island of rat sphingosine kinase-1 gene: tissue-dependent DNA methylation status and multiple alternative first exons. Genomics. 76: 117-125, 2001
S Tanaka, M Oda, Y Toyoshima, T Wakayama, M Tanaka, N Yoshida, N Hattori, J Ohgane, R Yanagimachi, K Shiota. Placentomegaly in cloned mouse concepti caused by expansion of the spongiotrophoblast layer. Biol Reprod. 65: 1813-1821, 2001
J Ohgane, J Aikawa, A Ogura, N Hattori, T Ogawa, K Shiota. Analysis of CpG islands of trophoblast giant cells by restriction landmark genomic scanning. Dev Genet. 22: 132-140, 1998
著書、総説
2011年以降
JN Muto T, K Iemitsu, T Watanabe, K Umeyama, J Ohgane, H Nagashima. Genetically engineered animal models for Marfan syndrome: challenges associated with the generation of pig models for diseases caused by haploinsufficiency. J Reprod Dev. 68, 233-237, 2022.
竹内健太、牧野智宏、新井良和、大鐘潤。ハプロ不全優性遺伝病発症の新たな視点-分子メカニズムとしてエピジェネティクスが関与する可能性-。明治大学農学部研究報告、65:95-103, 2016
2010年以前
K Ikegami, J Ohgane, S Tanaka, S Yagi, K Shiota. Interplay between DNA methylation, histone modification and chromatin remodeling in stem cells and during development. Int. J. Dev. Biol. 53: 203-214, 2009
J Ohgane, S Yagi, K Shiota. Epigenetics: the DNA methylation profile of tissue-dependent and differentially methylated regions in cells. Placenta. Suppl A, S29-35, 2007
大鐘潤、田中智、塩田邦郎。細胞特異的DNAメチル化プロフィール。蛋白質核酸酵素、
52:2177-2182、2007
K Nishino, J Ohgane, M Suzuki, N Hattori, K Shiota. Methylation in embryonic stem cells in vitro. Methods Mol. Biol. 329: 421-425, 2006
岩谷美沙、大鐘潤、塩田邦郎。発生・分化とエピジェネティクス。ゲノム医学 5:25-30、2005
大鐘潤、小田真由美、塩田邦郎。組織特異的メチル化と体細胞核移植クローン。エピジェネティクス(佐々木裕之編)、pp147-156、2004
J Ohgane, N Hattori, K Shiota. Analysis of tissue-specific methylation during development. Methods Mol. Biol. 289: 371-382, 2004.
招待講演
2011年以降
大鐘潤、新井良和。アリルごとのDNA メチル化状態に注目したエピジェネティクス研究の新たな試み。農芸化学分野におけるエピジェネティクスシンポジウム 日本農芸化学会2017年年会 京都 2017年3月
大鐘潤、新井良和、塩田邦郎。エピジェネティクス制御機構とエピゲノム変異検出。第28回環境ホルモン学会講演会 東京 2014年6月
大鐘潤、新井良和、塩田邦郎。ヘテロクロマチンを指標とした低レベル化学物質のエピ変異原性評価について。環境エピゲノミクス研究会第8回定例会 静岡 2013年11月
J Ohgane, Y Arai, K Shiota. Epigenetic assessment of environmental chemicals using mouse embryonic stem cells. AsiaCORD2013 Kobe, Japan 2013年4月
大鐘潤、新井良和、塩田邦郎。ヘテロクマチン形成を指標とした環境・人体に残存する低レベル化学物質のエピ変異原性評価に向けて。環境エピゲノミクス研究会第6回定例会 東京 2011年11月
2010年以前
J Ohgane, Locus-dependent DNA methylation changes in Sirt1 deficient mouse ES cells.6th NIBB-EMBL joint meeting of “Evolution of epigenetic regulation”, Heidelberg, Germany 2008年3月
J Ohgane, Evidence for simultaneous expression of a natural sense-antisense transcript pair from the ROSA26 locus: Implication for interference with transgene expression. 17th Lake Shirakaba Conference, Vedvaek, Denmark 2007年10月
J Ohgane. DNA methylation regulates Insulin gene expression. 16th Lake Shirakaba Conference, Grenada 2006年12月
大鐘潤、塩田邦郎。クローン動物の発生:DNAメチル化の意味。体細胞クローンから遺伝子変換動物への展望。第100回日本畜産学会 東京 2002年3月